전자의무기록 데이터 분석 접근법
Approach for Electronic Medical Record Data Analysis
Article information
Trans Abstract
As the healthcare environment is being digitalized and changed rapidly, research using medical big data is increasing. One of the most applicable data is electronic medical records which can provide a large amount of clinically practical meaning. Electronic medical data include patient's demographic information, laboratory test results, imaging and biosignal data. In this article, we provide support for a wide variety of researchers in their efforts to use electronic medical record data accurately and usefully in their work. From the basic concept of the research using electronic medical records to challenging aspects like data integration between multiple institutions are described. Also, examples of each type of data are covered; structured such as numeric data and unstructured such as images, biosignals and narrative text. Using these kinds of electronic medical records, analyses are processed by data cleansing, transforming, and reducing in order. Many kinds of variables such as the exposure and outcome of interest, covariate and the research design can be chosen during the preprocessing. As many machine-learning-based studies as well as epidemiologic-based studies have been conducted using electronic medical records, various research frameworks have been proposed. However, data quality management and data standardization for multi-center data analysis are still remaining as challenging tasks.
서 론
디지털화되고 급격하게 변화하는 보건의료 환경에서, 우리는 신속하면서도 정확하게 근거를 창출해낼 수 있는 능력을 갖추길 요구받고 있다. 특히 우리가 이전에 전혀 알지 못했던 Coronavirus disease 2019 (COVID-19)이 전세계적으로 유행하게 되면서, 새로운 바이러스의 특성을 알아내고 공유하고자 수많은 연구들이 쏟아져 나왔다. Nature지에 따르면 2020년도 한 해 동안에만 COVID-19과 관련된 연구가 전세계적으로 20만 건 이상 발표되었다고 한다[1]. 이렇게 빠른 기간 내에 특정 주제의 연구가 이 정도의 규모로 수행된 일은 역사상 처음일 것이다. 이처럼 많은 연구들이 단기간 내 수행될 수 있었던 배경에는 그 동안 다양한 보건의료데이터가 디지털화되어 빠르게 활용될 수 있는 환경이 크게 기여하였을 것이다.
보건의료데이터에서 가장 활발하게 저장, 관리, 활용되는 데이터는 각 병원의 전자의무기록 데이터와 보험 청구 자료(국민건강보험공단 혹은 건강보험심사평가원 자료)이다. 우리나라의 경우, 처방 및 검사 결과의 전산화는 2000년대 초반 이미 상당수 도입되어 있었으며[2], 2010년 이후 전자의무기록(electronic medical record, EMR)이 본격적으로 도입되기 시작하였다[3,4]. 2020년 한국보건의료정보원의 “보건의료정보화 실태조사”의 결과보고서에 따르면, 2020년도에는 대부분의 상급종합병원과 300병상의 종합병원에서는 전자의무기록을 사용하고 있다고 조사되었다[5]. 따라서, 처방 및 검사결과 정보는 약 20년 치의 데이터가 전산화되어 있으며, 의무기록 정보는 약 10년 치의 데이터가 전산화되어 있는 것이 현재 주요 병원들의 상황이라고 생각할 수 있다. 이렇게 전자의무기록데이터는 현재 충분히 많은 양이 쌓여 있을 뿐만 아니라, 보험 청구 자료 대비 자세한 환자 정보들이 기록되어 있다는 점에서, 빅데이터 시대 주요한 자료원으로 사용되고 있다.
2000년대 후반 이후 전자의무기록을 활용하여 발표된 연구들도 기하급수적으로 증가하였으며, 최근에는 전 세계적으로 매년 수천 편의 연구가 발표되고 있다[6] (Figure 1). 전자의무기록 데이터를 이용하여 연구를 할 경우, 다양한 장점과 단점이 있기에 이러한 특징을 잘 고려하여 분석을 진행해야 한다. 우선, 서두에 기술한 대로 이미 전산화된 형태로 데이터가 저장되어 있어서 신속하게 분석이 가능하다. 또한 추가적인 대상자 모집 등의 과정이 없기 때문에 분석 비용도 크게 필요로 하지 않는다. 그리고 무작위 대조연구에서는 윤리적인 이슈로 수행할 수 없는 다양한 조건의 분석의 경우도, 이미 분석 대상 조건이 기존 데이터에 존재한다면 분석에 포함시킬 수 있다. 하지만, 검사 치료 단계부터 의사의 개입이 반영되므로, 무작위성이 보장되지 못한다는 한계와 데이터 분석에 친화적이지 않은 원본 데이터의 한계로 인해, 이를 충분히 활용하는 데 어려움을 겪는 경우가 많이 있다.
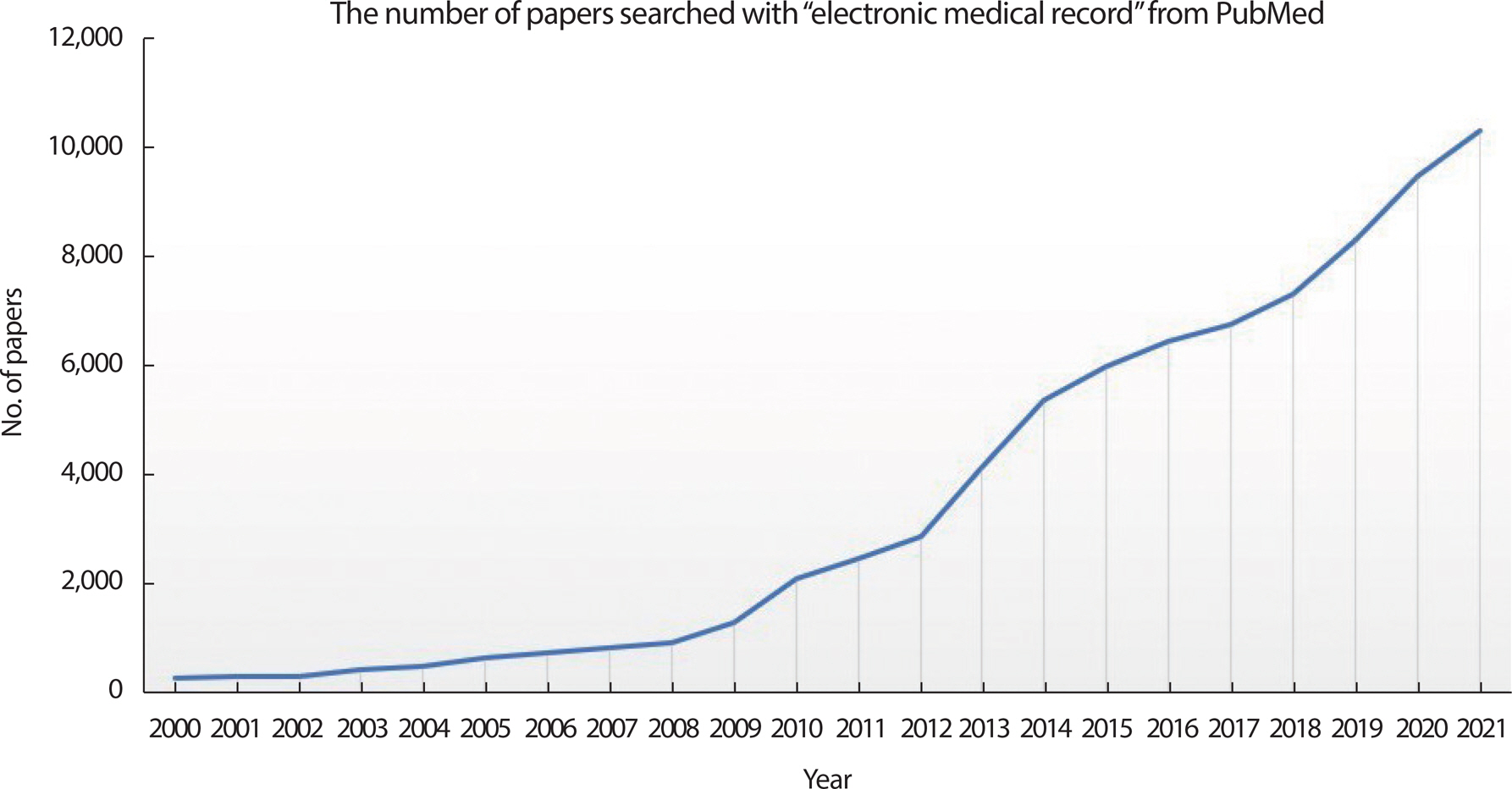
The trend of increasing the number of papers published using EMR. The number of papers searched from PubMed to electronic medical records (EMR) continues to increase.
이러한 한계를 극복하기 위해서는 의료 데이터에 대한 충분한 이해를 바탕으로 각 데이터별 적합한 전처리 과정을 진행해야 하며, 예측 가능한 비뚤림(bias)을 보정할 수 있는 연구 디자인을 활용해야 한다. 따라서, 본 글을 통해 보다 다양한 연구자들이 전자의무기록 데이터를 정확하고 유용하게 분석에 활용할 수 있도록 지원하고자 한다. 이를 위해, 전자의무기록 데이터의 분석에 대한 접근법을 정리하였다. 기본적인 용어와 개념, 데이터의 종류, 분석에 필요한 절차와 도구들, 그리고 도전적인 측면까지 다루고자 한다.
본 론
기본개념 및 용어
본격적인 이야기에 앞서 전자의무기록 데이터의 활용과 관련된 주요 개념들을 확인하고 넘어가고자 한다. 전자의무기록 데이터를 분석에 활용하는 일은 기본적으로 데이터의 ‘2차 활용’에 해당하게 된다[7]. ‘2차 활용’이라고 함은 데이터 수집의 본래의 목적과는 다르게 활용하는 상황을 말한다. 즉, 전자의무기록의 경우, 그 본래의 목적은 의료진의 진료 과정과 병원의 다양한 업무를 지원하고 법적인 의료 기록을 체계적으로 보관하기 위함이다.
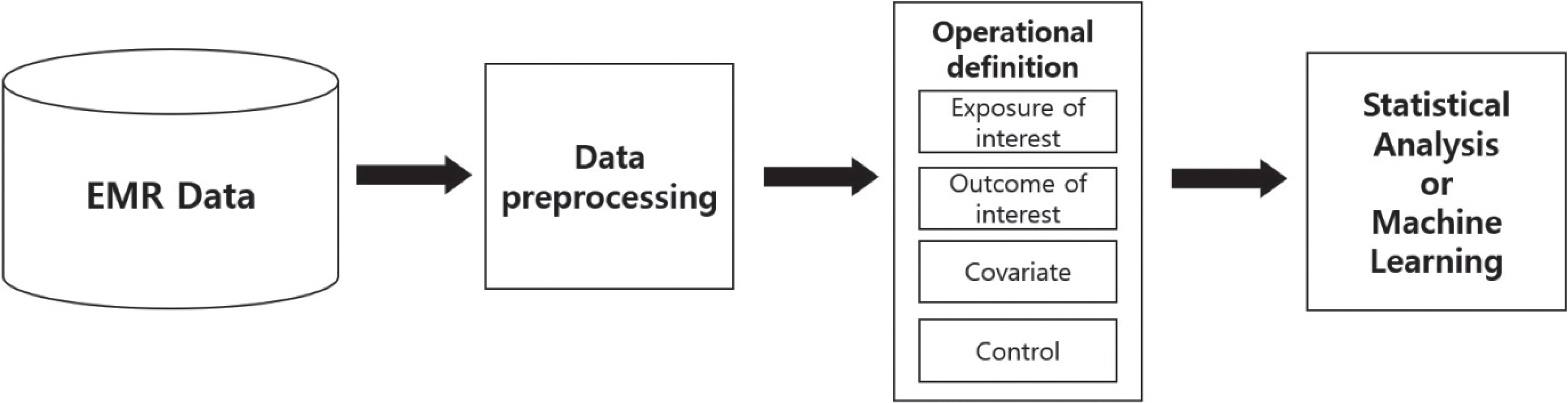
따라서, 우리는 전자의무기록 데이터를 분석하기 위해서는, 우리의 연구 가설에 맞게 기존의 데이터를 가공하는 과정이 필요하다. 일반적인 분석에서는 관심 노출, 관심 결과, 다양한 공변량을 기존 전자의무 기록 데이터에서 어떻게 정의할지를 결정해야 한다[7,8]. 이러한 과정을 ‘조작적 정의’라고 한다. 마지막으로, 전자의무기록 데이터의 분석은 기본적으로 ‘후향적 분석’이다. 후향적 분석이란, 분석하는 시점보다 과거에 이미 수집이 완료된 데이터를 활용하여 분석하는 방법을 말한다.
데이터 종류
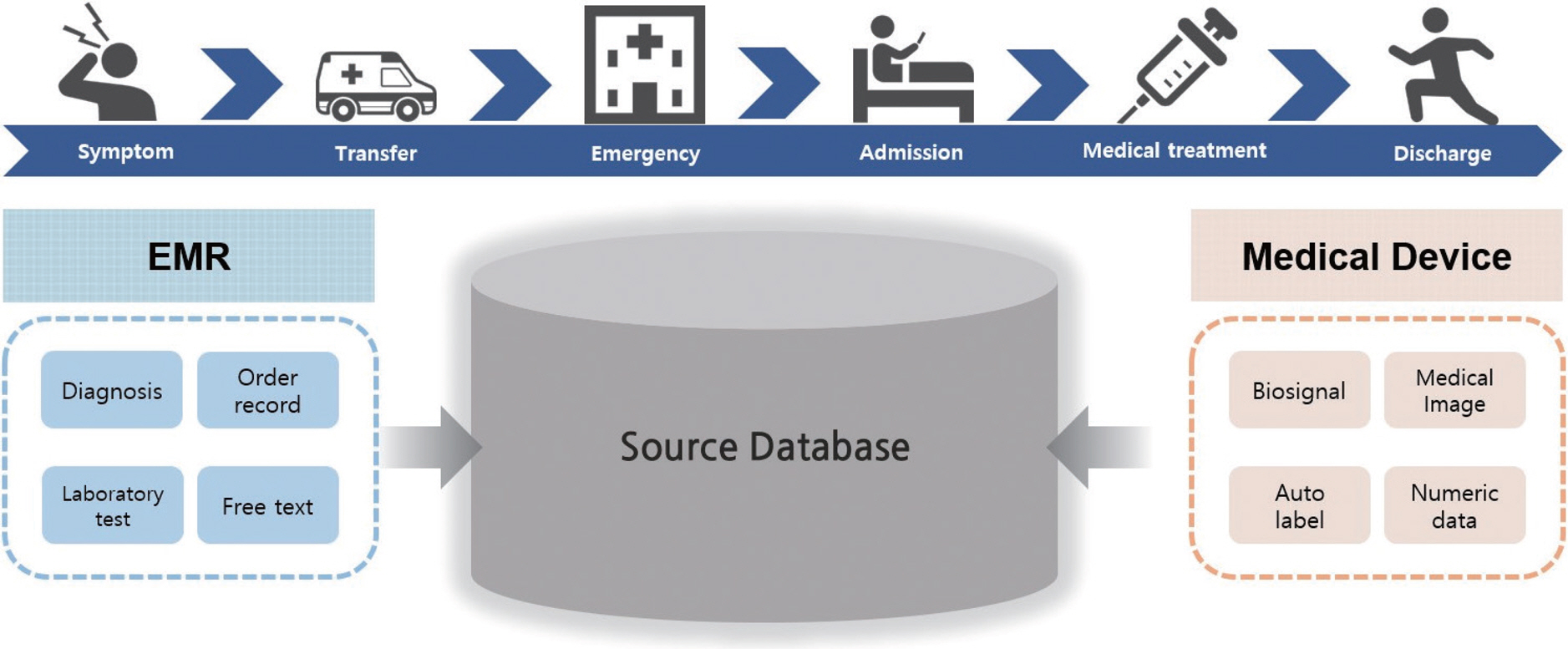
의료 데이터는 다양한 상황에서 다양한 종류의 데이터들이 생성되므로, 이를 분석하기 위해서는 전자의무기록에 저장되는 여러 데이터의 특성에 대한 이해가 선행되어야 한다(Figure 2).
정형 데이터
정형 데이터는 정해진 규칙에 따라 저장된 데이터를 말한다. 예를 들어, 진단명은 한국표준질병사인분류표의 분류 체계에 따라 기록이 되는 것이 일반적이다(예, ‘ E11.0’은 ‘혼수를 동반한 2형 당뇨병’을 의미). 검사, 약, 그리고 처치 처방들도 주로 각 기관에서 관리하는 처방 코드들을 사용하여 기록된다. 검사수치의 경우, 숫자형 데이터이거나 (+), (−) 등으로 미리 정해진 규칙 중 하나의 값을 갖는 범주형으로 표시된다. 환자 상태를 평가하기 위한 각종 척도들도 미리 정해진 기준에 따라 평가 후 점수화된다. 예를 들어 환자의 의식 수준을 평가하는 Glascow Coma Scale (GCS)은 눈을 뜨는 반응(1-4점), 언어반응(1-5점) 및 운동반응(1-6점)의 세 가지 평가항목의 반응을 점수화하여 그 합계 점수로 중증도에 따른 5단계의 의식 수준이 평가된다.
비정형 데이터
전자의무기록에 저장된 많은 양의 데이터는 비정형 데이터이다. 대표적인 예는 자유기술문으로, 현병력에 대한 진료 기록, 간호기록, 검사 판독문, 수술 기록 등 다양한 정보들이 자유기술문 형태로 저장되어 있다. 자유기술문에는 정형 데이터에서 기술하기 어려운 세부 내역이나, 처방의 사유 등 단순히 숫자나 범주로 보여지는 결과들 외에 자세한 정보들이 담겨 있다. 하지만, 현재까지의 분석 기술이 자유기술문을 처리하는 데에는 아직 충분하지 않은 실정이다. 특히, 전자의무기록 내의 자유기술문은 영문과 국문이 혼용되어 있으며, 전문적인 용어들이 사용되고 있고, 의도하지 않은 오타도 적지 않게 존재하며, 각 진료과별로 약어 등 언어 사용 패턴이 상이하다는 점이 기존의 일반적인 자연어처리 기술들을 바로 사용하는 데 장애 요소로 작용하고 있다. 또한 작성자 개인의 특성에 따라, 분량과 내용의 상세함이 달라서 정보의 양이 균일하지 않다는 한계도 존재한다.
영상 검사 보고서, 병리 검사 보고서 등 특정 보고서들은 시스템에 의해 형식이 규정되지는 않으나, 작성자가 일정한 규칙을 갖고 작성하는 경우가 대부분이어서 반정형 데이터(semi-structured data)로 불리기도 한다. 이런 경우, 정규표현식과 같은 방법을 이용하여 필요한 정보를 추출하는 일이 가능하다[9]. 하지만, 시스템에 의해 형식이 규정되지 않았기에, 여전히 다양한 예외들이 존재할 가능성이 높고, 시기에 따라 형식이 바뀌거나, 의료진의 변경에 따라 형식이 바뀌는 경우들이 종종 있어, 활용에 제한이 따른다.
영상 이미지의 경우, 형식 자체는 정해진 이미지의 크기로 규격화되어 있지만, 우리가 분석에 포함하고자 하는 관심 정보(즉, 관심 노출, 관심 결과, 혹은 공변량)들이 이미지 내 정해지지 않은 형태로 존재하기 때문에 비정형 데이터로 분류하는 것이 일반적이다. 최근 디지털 병리 솔루션의 도입이 진행되어, 병리 슬라이드도 영상 이미지로 저장되는 곳이 늘어나고 있는 한편, 다양한 보고서들이 데이터화되기 힘든 스캔본 형식으로 저장되어 있는 경우도 다수 존재한다.
또한 심전도, 호흡패턴, 동맥압 파형 등 다양한 생체신호 데이터들도 병원에서 수집되는 대표적인 비정형 데이터 중 하나이다. 파형 생체신호는 중환자실 입실 기간 또는 입원 기간 동안 체외 부착 또는 체내 삽입된 수집 기기로부터 연속적으로 수집된 수치를 저장하고 있으며, 다른 형태의 데이터에 비해 상대적으로 높은 수집 빈도를 갖고 있다. 심전도 데이터는 심장의 상태를 직접적으로 대변하는 정보들뿐만 아니라, 전신의 순환 상태 등 다양한 임상적 정보들이 비정형적 형태로 존재한다[10]. 심전도뿐만 아니라, 호흡패턴, 동맥압 파형 등에도 주요한 임상적 이벤트를 탐지하거나 예측하는 데 유용한 정보들이 복잡하게 숨어 있다[11,12].
각 데이터 종류로 분석한 예시
상기 기술한 다양한 종류의 데이터로 분석한 여러 연구들이 발표되고 있다.
Tomasev et al. [13]에서는 1,243개의 의료기관에서 수집하여 취합된 약 70만 명의 전자의무기록에서 진단 코드 (International Classification of Diseases, 9th edition, ICD-9), Current Procedural Terminology (CPT) 코드, 각종 검사 수치 데이터, 처방기록 등의 정형 데이터를 추출하여 급성 신부전을 연속적으로 예측하는 인공지능 모델을 개발하여 발표하였다. 해당 연구에서는 심층 신경망(deep neural network)을 활용하여 이러한 정형 데이터에서 특징을 추출하였으며, 입원 환자들 중에서는 급성 신부전 에피소드의 55.8%, 투석 치료가 필요한 환자들 중에서는 90.2%의 에피소드를 예측하였다.
Gulshan et al. [14]에서는 128,175장의 망막 안저 사진 데이터를 활용하여 당뇨병성 망막증을 탐지하는 인공지능 모델을 개발하여 발표하였다[14]. 해당 연구에서는 상기 언급한 비정형 데이터(망막 안저 사진)만을 활용하였고, 합성곱 신경망(convolutional neural network)을 활용하여 이러한 이미지 데이터로부터 특징을 추출하였으며 area under the receiver operating curve (AUROC) 0.990의 성능으로 당뇨병성 망막증을 탐지하였다.
Raghunath et al. [15]에서는 표준 12 유도 심전도(standard 12 lead electrocardiogram) 데이터를 활용하여 1년 이내 사망을 예측하는 인공지능 모델을 개발하여 발표하였다. 해당 연구에서는 상기 언급한 비정형 데이터(표준 12 유도 심전도) 데이터만을 활용하였고, 합성곱 신경망을 활용하여 이러한 심전도 데이터로부터 특징을 추출하였으며, AUROC 0.88의 성능으로 1년 이내 사망을 예측하였다.
또한, 다양한 종류의 데이터를 함께 활용한 연구도 발표되고 있다. Goh et al. [16]에서는 전자의무기록의 각종 검사 수치, 처방 기록 등의 정형 데이터와 자유기술문 형태의 임상 노트 데이터를 함께 활용하여 패혈증을 예측하는 인공지능 모델을 개발하여 발표하였다. 해당 연구에서는 자유기술문 데이터로부터 특징을 추출하기 위해 잠재 디리클레 할당(latent dirichlet allocation, LDA)라는 방법을 활용하였고, 심층 신경망 구조를 활용하여 AUROC 0.94의 성능으로 패혈증을 발생 12시간 전에 예측하였다.
전자의무기록 데이터에 대한 접근
전자의무기록 데이터 분석을 위한 첫 단계는 데이터에 대한 접근 권한을 얻는 것이다. 가장 일반적인 방법은 본인이 근무하는 병원에서 제공하는 임상 데이터 웨어하우스(clinical data warehouse, CDW)를 이용하여 데이터를 확보하는 것이다. CDW 시스템이 갖춰지지 않은 경우, 각 병원의 전산 혹은 의무기록 업무를 담당하는 부서를 통해 데이터를 확보할 수 있을 것이다.
데이터에 대한 접근 권한을 얻기 위해서는 기관생명윤리위원회의 승인을 얻어야 하며, 가명처리 및 외부 반출을 위해서는 데이터심의위원회의 승인을 얻어야 한다. 데이터심의위원회의 경우, 2021년에 개정된 데이터 3법에 따라 보건복지부에서 발표한 ‘2021년 보건의료 데이터 활용 가이드라인’을 준수하고자 주요 병원들에서 구성 및 운영되고 있다[17].
공개된 전자의무기록 데이터베이스 활용
병원의 전자의무기록 데이터에 직접적인 접근이 어려운 연구자의 경우, 두 가지 방법이 있다. 분석하고자 하는 병원의 연구자와 공동 연구를 수행하거나, 공개된 데이터를 이용하는 것이다. 공개된 전자의무기록 데이터베이스가 다수 존재하지만, 대부분은 환자당 데이터의 기록 빈도가 높은 중환자실 데이터이다. 대표적으로, Medical Information Mart for Intensive Care III 및 IV (MIMIC-III or MIMIC-IV) 데이터베이스는 미국의 the Beth Israel Deaconess Medical Center 중환자실에 내원한 성인 및 신생아 환자의 데이터를 전 세계 연구자들에게 무료로 공개한 데이터베이스이다[18]. MIMIC-III/IV는 인구학적 정보, 진단, 처방 및 검사 정보, 임상 노트 등과 같은 전자의무기록 데이터를 포함하고 있고 뿐만 아니라 중환자실에서 모니터링 되고 있는 심전도 등의 파형 데이터, 흉부 x-ray 데이터 등의 비정형 데이터도 포함한다. MIMIC-III/IV는 다양한 중환자 관련 연구를 위해 활용되어 왔고 새로운 임상 의사결정 알고리즘 개발에 기여하고 있다. 한국 버전의 K-MIMIC 데이터도 수년 내에 공개될 예정이다. 또 다른 공개 중환자 전자의무기록 데이터베이스로는 eICU 데이터베이스가 있다[19]. 20만 건 이상의 중환자실 방문에 대한 인구학적 정보, 진단, 처방, 검사 및 활력징후 데이터를 포함하지만, 임상 노트와 파형 데이터는 포함하고 있지 않다. MIMIC-III 데이터베이스와 동일한 연구 그룹에서 관리되고 있다. 이러한 다양한 공개 데이터베이스를 활용하여 하나의 병원 혹은 데이터베이스에서 개발된 분석 모델을 다른 데이터베이스로 외부 검증하는 형태의 연구도 가능하다.
데이터 전처리
데이터에 대한 접근이 가능해지면, 데이터를 전처리 과정이 선행되어야 한다(Figure 3). ‘데이터 전처리’는 원본 데이터를 통계 분석이나 기계학습 모델 개발에 적합하도록 데이터를 가공하는 일련의 과정을 말한다[7]. 의료 데이터 분석을 처음 시도한다면, 여러 실습 교재에서 제공되는 정제된 예시 데이터와 달리 실제 원시 데이터가 낯설게 느껴질 수 있다. 이렇게 정제되지 않은 데이터를 분석에 적합하게 전처리하는 과정이 없다면 편향된 분석 결과로 이어질 수 있다. 따라서 전처리의 수준에 따라 분석의 정확도가 결정되며, 전체 데이터 분석에 소요되는 시간과 노력 중 약 70-80%가 데이터 전처리 과정에 쓰이는 것이 일반적이다.
특히, 전자의무기록데이터와 같은 실제 세계의 데이터(real world data, RWD)에는 치료 프로세스에 따른 특유의 오류 패턴이 있어, 단순한 이론적인 내용으로만 온전한 전처리를 진행하기 어렵고, 다양한 의료 데이터 분석 경험을 통해 노하우를 쌓아야 한다. 또한 동일한 데이터에 대해서도 분석하고자 하는 가설 및 설정에 따라 그 처리 방식이 달라질 수 있기 때문에, 데이터와 분석 방법에 대한 깊은 이해를 필요로 한다.
데이터 전처리는 다음의 단계로 진행된다. 첫 번째는 원본 데이터의 수집과정에서 발생된 오류들을 바로잡는 ‘데이터 정제’ 단계이다. 결측치, 잡음, 이상치, 불일치 데이터를 처리한다. ‘결측치’는 관측되지 않은 정보로, 결측값이 많은 변수의 경우(예: 50% 이상), 해당 변수를 더 이상 분석에 포함하는 것은 옳지 않다[7]. 그렇지 않다면 다양한 대치 방법(평균값과 중앙값, 선형 회귀, 혹은 다중 대치법 등)을 통해 결측치를 다른 값으로 대치하여 분석을 수행할 수 있다. ‘잡음’은 의도치 않은 간섭(예: 피수집자의 움직임, 수집기기의 작동불량 등)에 의해 발생하는 속성값의 오류로 정의되며, 측정 과정에서 발생하는 문제로 잘못된 데이터나 왜곡된 데이터를 포함한다[20]. 이와 달리 ‘이상치’는 보통 관측된 데이터의 범위에서 많이 벗어난 값으로, 실제 관심 있는 데이터지만 비정상적으로 보일 수 있다. 이상치는 결과에 영향을 줄 수 있기 때문에 이를 파악하여 적절하게 처리해야 한다[21].
두 번째 단계는 통계적 분석 및 기계학습에 유용한 형태로 데이터를 변형하는 ‘데이터 변환’ 과정이다. 분석에 더 적합한 형식 및 척도를 갖도록 정규화 하거나, 비슷한 의미를 갖는 세부 정보를 통합하는 과정이 포함된다. 분석 방법 및 사용하는 데이터의 종류에 따라 필요한 데이터의 형태 또한 달라진다. 정형 데이터를 활용한 통계적 분석 및 기계학습에는 표 형식으로 정렬된 데이터가 일반적으로 사용되며, 이미지 등 비정형 데이터를 사용한 기계학습에는 데이터를 다차원의 배열로 변형하여 사용하는 것이 일반적이다.
마지막 단계는 데이터 분석을 명확하게 진행하기 위해 중복되거나 세분화된 정보를 통합하는 ‘데이터 축소’ 단계이다. 진단코드 체계를 이용하는 등 전문적 지식에 기반한 규칙대로 데이터를 축소시킬 수도 있고, 수학적 모델 혹은 기계학습 방법 등을 활용하여 데이터를 축소시킬 수 있다. 모든 분석에서 반드시 수행되어야 하는 단계는 아니지만, 전자의무기록 데이터와 같이 대규모의 다차원 데이터를 다루는 경우에는 분석을 용이하게 하기 위해 이 과정을 필요로 하게 된다[22].
데이터 분석 설계
역학적 연구 디자인 기반의 분석 설계 방법
전자의무기록을 활용한 데이터 분석을 위해서는 다양한 역학적 연구 디자인 중 본인이 의도하는 측면으로의 연구 디자인 설계가 필수적이며, 이는 향후 연구 결과에 영향을 끼치기도 한다. 전자의무기록의 대표적인 분석 디자인인 후향적 분석은 크게 관심 노출에 노출된 그룹과 그의 대조군을 선정하고, 노출 이후에 발생하는 관심 결과의 발생 빈도를 살펴보는 후향적 코호트 연구, 혹은 관심 결과가 발생한 그룹과 그의 대조군을 선정한 후 그 이전에 있었던 노출들의 빈도를 조사하는 환자-대조군 연구 방법이 대표적이다[23]. 전통적인 역학의 관점에서 바라보면, 후향적 코호트 연구가 노출과 시간에 따른 시간의 영향을 살펴볼 수 있다는 장점이 있고, 관심 노출에 노출된 환자의 수가 적을 경우 활용하기 적합하다는 특징이 있다. 반면, 관심 결과 발생이 적을 경우에는 미리 관심 결과가 발생한 환자들을 선별하고 분석하는 환자-대조군 연구가 적합할 수 있다. 하지만, 데이터를 모두 활용할 수 있는 전자의무기록 데이터 분석 상황에서는, 데이터의 수집 현황에 따라 적절한 연구 디자인이 적용되어야 한다. 우리가 설정한 관심 결과가 발생할 때 발생 상황과 발생 시점이 적절히 확인될 수 있는 경우 관심 노출과 관심 결과 간의 시간의 영향을 분석할 수 있기 때문에 후향적 코호트 연구가 적절할 것이다. 반면, 특정 시점이 적절하게 확인되기 힘든 경우는 환자-대조군 연구가 적절할 것이다. 예를 들어, 부정맥의 발생을 관심 결과로 하고 이와 관련된 인자들을 찾고자 하는 경우, 심전도의 변화가 지속적으로 관찰되고 기록되어야 한다. 그러나 심전도의 경우 보통 1년에 1회 정도 측정되는 경우가 대부분이므로 심전도의 변화를 지속적으로 관찰하기 어렵다[24]. 이 경우, 부정맥 발생의 시점을 정확하게 추정할 수 없으므로 환자-대조군 연구 방법을 활용하는 것이 적절할 것이다.
기계 학습 기반의 분석 설계 방법
최근에는 전자의무기록을 이용한 연구에서 전통적인 역학적 연구 설계에 기반을 두지 않고, 다양한 기계 학습 방법을 적용하여, 데이터 내 존재하는 유의미한 패턴을 찾고자 하는 시도들이 증가하고 있다[12]. 이 경우, 관심 결과와 연관성이 있는 인자들의 위험도 혹은 연관성을 탐색하기 보다는 관심 결과의 발생 위험을 예측하는 등 임상 현장에서 활용될 수 있는 인공지능 모델을 만드는 일에 더 초점을 맞추게 된다. 따라서, 실제 임상 환경에서 적용될 상황을 반영하여, 관심 결과의 예측을 위해 패턴을 학습해야 하는 관찰 기간 및 효용성을 위해 필요한 예측 구간에 따라 다음과 같이 다양한 연구 설계를 설정할 수 있다: 발병 전 고정된 기간, 슬라이딩 윈도우(sliding window), 동적으로 배정 후 슬라이딩 윈도우(sliding window with dynamic allocation), 임상적 요구 시점, 순차적, 결과 발생 당시, 발병 시점까지의 무작위 기간[25].
전자의무기록 데이터 분석 수행
전처리가 완료된 데이터를 연구 설계에 맞게 준비하였다면, 그 다음으로는 실제 분석을 수행하는 일이 남았다. 분석을 위해서는 다양한 접근 방법이 고려될 수 있다. 생존 여부와 같은 이진 결과에 대한 요인을 분석하고자 할 경우, 로지스틱 회귀분석이나 콕스(Cox) 회귀분석 등이 사용될 수 있을 것이다. 또한 최근에는 다양한 기계학습 모델들을 활용하여 특정 집단들 간의 유용한 패턴 차이를 밝혀내는 연구들이 많이 발표되고 있다. 자세한 분석 방법에 대해서는 본 글의 범위를 벗어나므로 자세히 언급하지는 않도록 하겠다. 분석 기법 자체에 대해서는 다양한 선행 연구들에서 쉽게 확인할 수 있을 것이다.
필요 프로그래밍 도구
전 세계의 데이터 과학자들을 대상으로 한 설문조사에 따르면, 데이터 과학을 하는 데에 있어서 가장 많이 쓰이는 프로그래밍 언어는 Python (87%), SQL (44.3%), R (31%)이었다[26]. Python과 R은 모두 사용자가 통계 분석 및 기계 학습을 포함하여 데이터 과학의 모든 기술을 수행할 수 있도록 하는 무료 패키지들이 개발되어 있지만, 상대적으로 Python은 기계학습에 특화된 패키지들이, R에는 통계 분석에 집중된 패키지들이 배포되어 있다. 또한 두 언어 모두 데이터의 시각화에 매우 탁월하다. 반면에 SQL은 관계형 데이터베이스에서 데이터를 검색 및 조작하는 질의(query)를 수행하기 위해 특별히 설계된 데이터베이스 언어이다.
도전들
다기관 분석
데이터 분석 대상에 해당하는 충분한 환자 수를 확보하기 위해, 그리고 개발된 분석 모델을 다양한 환경에서 신뢰성 및 일반화 가능성한지 평가하기 위해서는 단일 기관 분석보다는 다기관 분석이 필요하다. 앞서 언급한 것처럼, 상당수의 의료기관에서, 특히 상급종합병원에서는 이미 대부분 전자의무기록이 도입된 상태이지만, 각 의료기관마다 방문하는 환자의 특성이 다를 수 있고, 시행되는 임상 프로세스가 상이할 수 있고, 전자의무기록 데이터베이스의 형식 및 체계가 다른 경우가 대부분이다. 따라서 다기관 분석을 위해서는 수동적으로 필요한 변수를 선정하고 정제하는 과정을 거치거나, 사전에 정해진 형식으로 통일하는 표준화 과정이 필요하다. 다기관 분석의 예로 우리나라 3차 병원 두 곳(아주대학교병원, 가천대학교 길병원)의 방문 기록과 검체 검사 결과를 활용하여 Z-정규화를 기반으로 한 Subgroup-adjusted normalization method (SAN) 기법을 개발한 연구가 있으며, 이를 통해 특성이 다른 두 기관 간의 데이터를 비교 및 정규화함으로써 다기관 분석이 가능하게 하였다[27].
표준화
우리나라 병원의 전자의무기록 활용 비율이 지속적으로 증가함에 따라, 앞서 서술한 다기관 분석을 위해서는 각 병원의 전자의무기록의 형식 표준화가 필수적이며 이것이 연구를 위한 중요한 도전 과제 중 하나로 여겨지고 있다. 표준화란 각 기관에서 사용하고 있는 데이터의 명칭, 형식 등을 하나로 통일하여 대규모 분석 연구가 가능하도록 하는 중요 프로세스이다. 예를 들어, A병원에서는 측정단위로 mg, B병원에서는 측정 단위로 g을 사용한다거나, 하나의 병원에서 생체신호 측정을 위해 두 제조사의 기기를 사용하여 측정 단위나 Hz, 시간 등이 상이한 경우 데이터의 표준화가 필수적이다. 3차 병원의 전자의무기록을 다기관 연구를 위한 표준화된 데이터 모델인 Observational Medical Outcomes Partnership-Common Data Model (OMOP-CDM) 4.0 버전 형식으로 통일시키기 위해 전자의무기록의 날짜 형식을 YYYY-MM-DD에서 DD-MM-YY로 변환하는 등의 표준화를 진행하여 다기관 연구를 위한 기반을 마련하기도 하였다[28]. 이처럼 원활한 다기관 대규모 분석 연구를 위해서는 공통의 표준화된 “약속”을 제정하는 것이 중요하다. 또한, 전자의무기록의 표준화된 품질을 확보하기 위하여 우리나라에서는 2020년부터 전자의무기록 인증제(EMR인증제)를 실시하여 시스템의 상호 호환성 및 표준화된 품질을 보장하고자 하고 있다.
결 론
디지털화된 병원 환경과 복잡한 데이터를 분석할 수 있도록 지원하는 여러 분석 방법의 발달로, 전자의무기록 데이터 내 저장된 대규모의 의료 데이터를 활발하게 분석할 수 있는 환경이 조성되었다. 또한, 많은 수의 연구 결과, 특히 코로나 시대에 필요한 근거를 빠르게 생산한 경험들은 전자의무기록 데이터 분석의 중요성을 높이고 있다. 현재는 진료라는 1차 목적에 최적화된 데이터들이 전자의무기록 내에 기록되고 있지만, 의료 데이터 분석의 중요성이 널리 인식되고 있음에 따라, 보다 데이터 활용에 적합한 형태로 발전해 나갈 것으로 기대되며, 결국 현재의 한계를 넘어 더욱 다양하고 중요한 근거들을 생산하는 중요한 데이터 소스로 널리 활용될 것이다.